2022
05-13
05-13
如何利用WGCNA分析转录组数据?
 一、WGCNA分析简介随着转录组测序价格下降,进行测序的样本数也在逐渐增多,WGCNA(weighted gene co-expression networkanalysis,权重基因共表达网络分析)这种适合大样本的分析方法,在疾病以及其他性状与基因关联分析等方面应用越来越广泛。WGCNA最大的优势在于,能将多个样本中成千上万的基因,根据它们的表达模式划分到数个至数十个模块当中,然后以模块...阅... 阅 读 全 部 >
一、WGCNA分析简介随着转录组测序价格下降,进行测序的样本数也在逐渐增多,WGCNA(weighted gene co-expression networkanalysis,权重基因共表达网络分析)这种适合大样本的分析方法,在疾病以及其他性状与基因关联分析等方面应用越来越广泛。WGCNA最大的优势在于,能将多个样本中成千上万的基因,根据它们的表达模式划分到数个至数十个模块当中,然后以模块...阅... 阅 读 全 部 >
 研究背景雌激素是一种甾体激素,对雌性繁殖有广泛的影响。在生殖期,雌激素的合成主要在卵巢卵泡的体细胞(卵泡膜细胞,TCs和颗粒细胞,GCs)中。然而在哺乳动物和鸟类中其合成位置并不相同。在哺乳动物中,TCs从头产生雄激素,GCs进行雌二醇转化。然而由于未知的原因,在鸟类中TCs产生睾酮和雌二醇(一种雌激素)。CYP19A1编码芳香酶,是负责雌激素的生物合成的关键步骤。哺乳动物牛中,雌...阅读全文&...
研究背景雌激素是一种甾体激素,对雌性繁殖有广泛的影响。在生殖期,雌激素的合成主要在卵巢卵泡的体细胞(卵泡膜细胞,TCs和颗粒细胞,GCs)中。然而在哺乳动物和鸟类中其合成位置并不相同。在哺乳动物中,TCs从头产生雄激素,GCs进行雌二醇转化。然而由于未知的原因,在鸟类中TCs产生睾酮和雌二醇(一种雌激素)。CYP19A1编码芳香酶,是负责雌激素的生物合成的关键步骤。哺乳动物牛中,雌...阅读全文&...  COG是Clusters of Orthologous Groups of proteins的缩写(http://www.ncbi.nlm.nih.gov/COG/)。COG是在对已完成基因组测序的物种的蛋白质序列进行相互比较的基础上构建的,COG数据库选取的物种包括各个主要的系统进化谱系。每个COG家族至少由来自3个系统进化谱系的物种的蛋白所组成,所以一个COG对应于一个古老的保守结构域。构成每...
COG是Clusters of Orthologous Groups of proteins的缩写(http://www.ncbi.nlm.nih.gov/COG/)。COG是在对已完成基因组测序的物种的蛋白质序列进行相互比较的基础上构建的,COG数据库选取的物种包括各个主要的系统进化谱系。每个COG家族至少由来自3个系统进化谱系的物种的蛋白所组成,所以一个COG对应于一个古老的保守结构域。构成每... 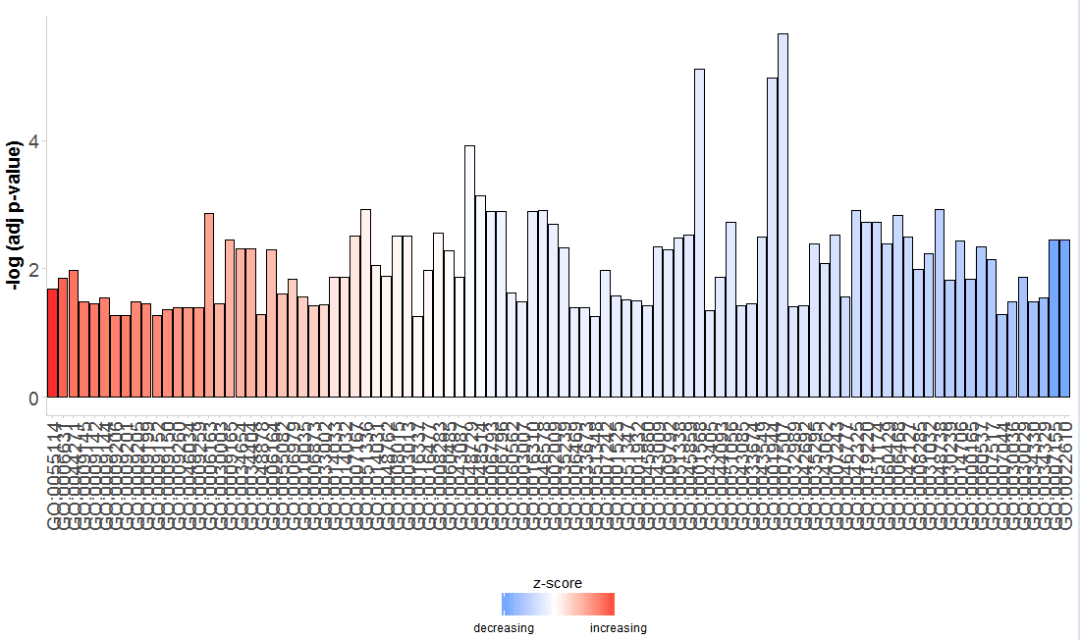 GO分析相信是所有小伙伴都需要用到的,那么,我们如何使用R中的GOplot包绘制一张完美的GO富集分析图呢?首先,我们打开R,用以下命令进行 G O p l o t 包的安装install.packages('GOplot')#安装GOplot包安装完后,我们将R包及分析好的GO数据导入library(GOplot)#导入R包data(EC) #导入数据head(EC$david)#...阅读全文...
GO分析相信是所有小伙伴都需要用到的,那么,我们如何使用R中的GOplot包绘制一张完美的GO富集分析图呢?首先,我们打开R,用以下命令进行 G O p l o t 包的安装install.packages('GOplot')#安装GOplot包安装完后,我们将R包及分析好的GO数据导入library(GOplot)#导入R包data(EC) #导入数据head(EC$david)#...阅读全文... 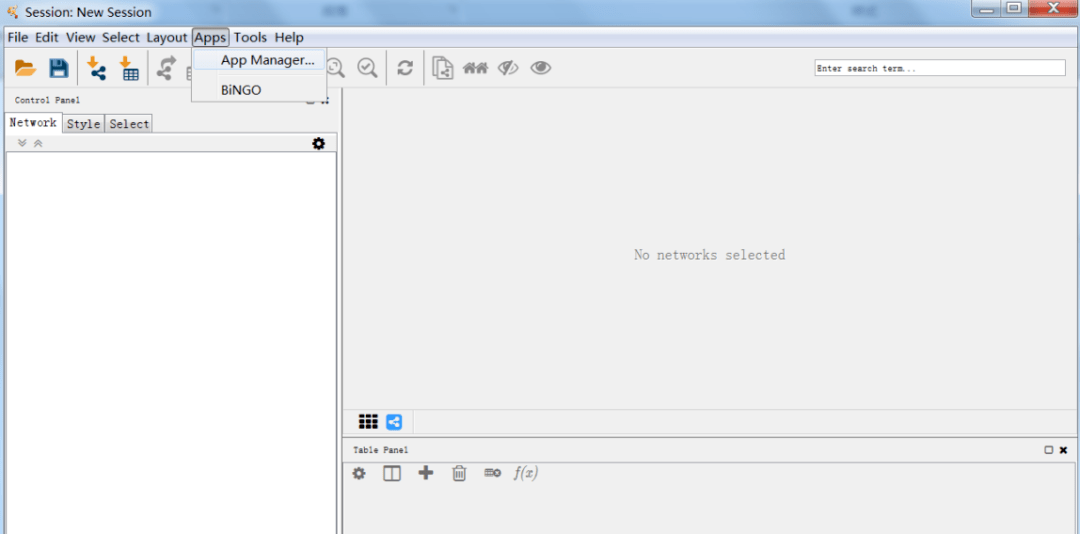 Cytoscape是一个优秀的生物软件平台,可以完成很多的生物学分析,我们可以使用cytoscape中的bingo插件,完成模式物种的GO富集分析。首先,打开cytoscape,安装bingo插件运行bingo输入保存名称输入差异基因输入检验方式与阈值输入要聚类的条目选择物种保存运行,得到富集结果可以看到,颜色越深,代表越显著,圆圈越大,代表富集到的基因越多颜色表示的数值可以将网络图导出...阅读...
Cytoscape是一个优秀的生物软件平台,可以完成很多的生物学分析,我们可以使用cytoscape中的bingo插件,完成模式物种的GO富集分析。首先,打开cytoscape,安装bingo插件运行bingo输入保存名称输入差异基因输入检验方式与阈值输入要聚类的条目选择物种保存运行,得到富集结果可以看到,颜色越深,代表越显著,圆圈越大,代表富集到的基因越多颜色表示的数值可以将网络图导出...阅读... 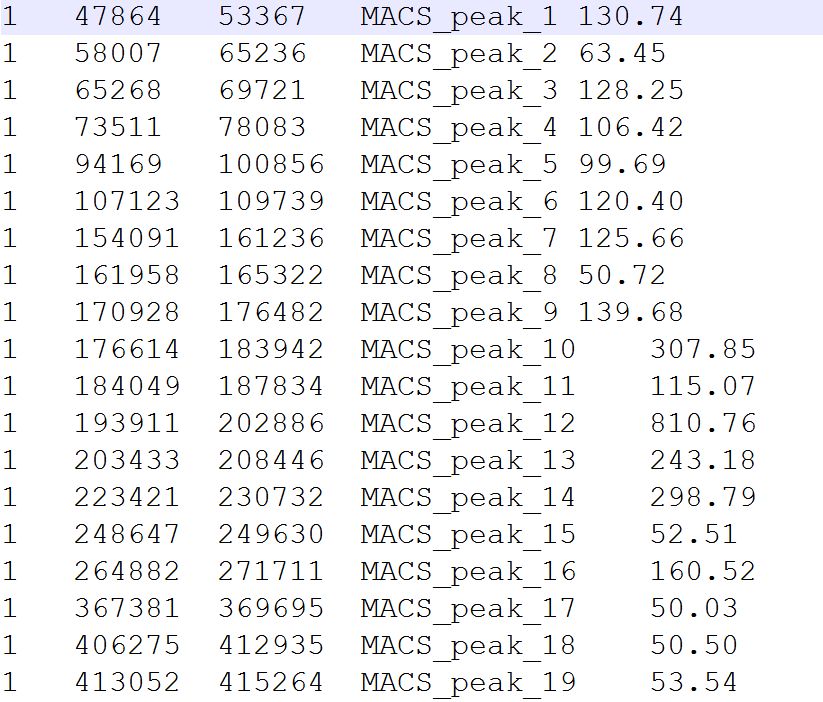 ChIP-seq主要用来研究蛋白质和DNA的相互作用,ChIPseeker 可以用来对ChIP-seq数据进行注释与可视化,下面我们就来介绍一下如何用ChIPseeker对chip-seq数据进行可视化操作。操作步骤把所有sample_peaks文件放在工作路径下,格式为#安装程序#source("http://bioconductor.org/biocLite.R") #biocL...阅读全文...
ChIP-seq主要用来研究蛋白质和DNA的相互作用,ChIPseeker 可以用来对ChIP-seq数据进行注释与可视化,下面我们就来介绍一下如何用ChIPseeker对chip-seq数据进行可视化操作。操作步骤把所有sample_peaks文件放在工作路径下,格式为#安装程序#source("http://bioconductor.org/biocLite.R") #biocL...阅读全文... 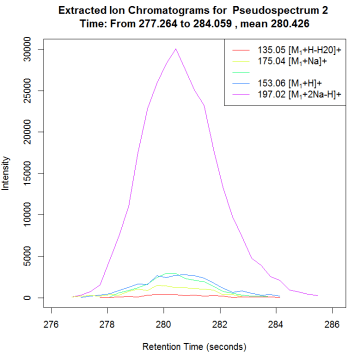 今天分享的是使用CAMERA对代谢物加和峰进行注释。下面我们就来介绍一下具体方法。操作步骤#载入R包library(CAMERA)#单个样品file <- system.file('mzdata/MM14.mzdata', package = "CAMERA") xs <- xcmsSet(file,method="centWave",ppm=30,peakw...阅读全文>&g...
今天分享的是使用CAMERA对代谢物加和峰进行注释。下面我们就来介绍一下具体方法。操作步骤#载入R包library(CAMERA)#单个样品file <- system.file('mzdata/MM14.mzdata', package = "CAMERA") xs <- xcmsSet(file,method="centWave",ppm=30,peakw...阅读全文>&g... 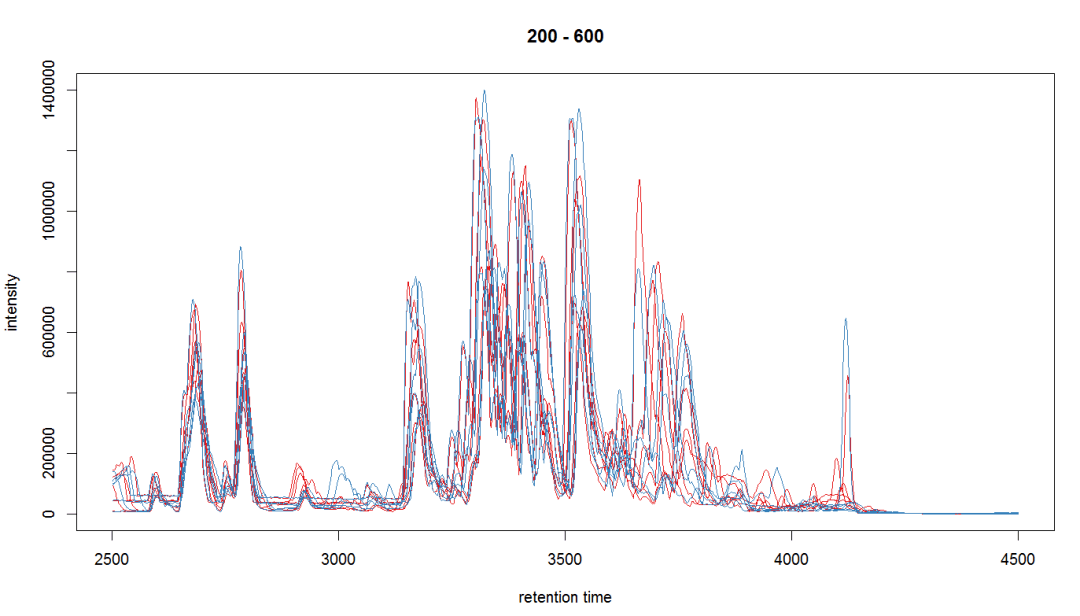 xcms是基于R语言设计的程序包(R package),可以用分析代谢组数据等。下面我们就来介绍一下使用方法。操作步骤示例数据是fatty acid amide hydrolase (FAAH) 基因敲除鼠的脊柱LC-MS,六个基因敲除个体,六个野生型,使用的是centroid mode ,正离子模式,200-600 m/z ,2500-4500 seconds。source("htt...阅读全...
xcms是基于R语言设计的程序包(R package),可以用分析代谢组数据等。下面我们就来介绍一下使用方法。操作步骤示例数据是fatty acid amide hydrolase (FAAH) 基因敲除鼠的脊柱LC-MS,六个基因敲除个体,六个野生型,使用的是centroid mode ,正离子模式,200-600 m/z ,2500-4500 seconds。source("htt...阅读全... 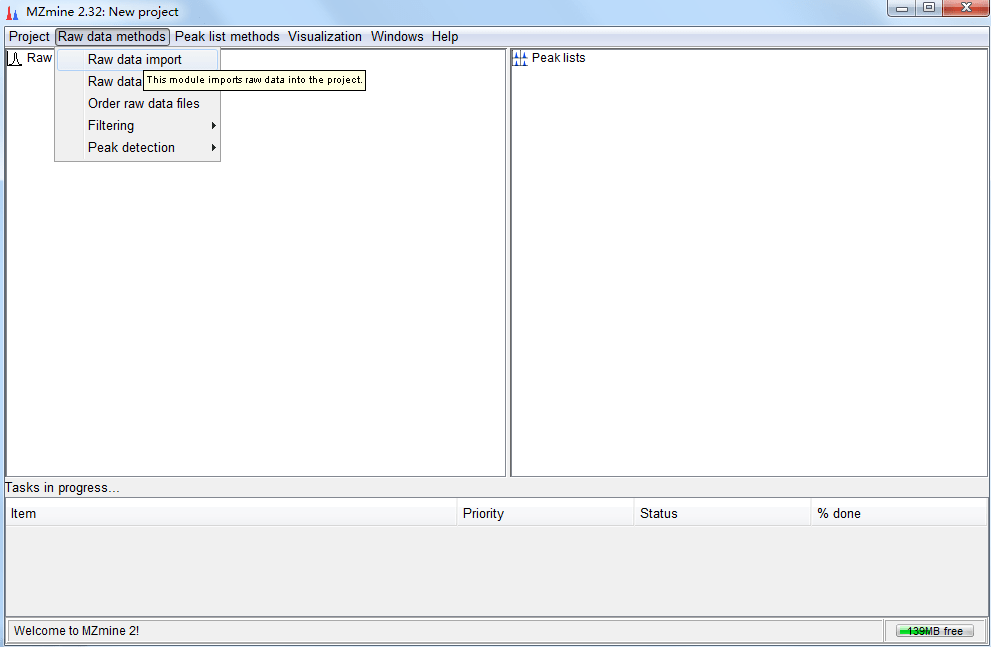 提到代谢组学峰比对,不得不提到一款老牌的比对软件,它就是MZmine2。它发表于2010年,前身是MZmine,发表于2006年,可谓是十分经典的一款代谢组学峰比对软件。MZmine 2的功能有很多,它是一款基于JAVA开发的开源软件,可以用于处理代谢组学profile数据,并且具有可视化和很多的分析功能。MZmine 2的一部分功能基于R,所以首先需要安装R环境,并且安装一些依赖包:instal...
提到代谢组学峰比对,不得不提到一款老牌的比对软件,它就是MZmine2。它发表于2010年,前身是MZmine,发表于2006年,可谓是十分经典的一款代谢组学峰比对软件。MZmine 2的功能有很多,它是一款基于JAVA开发的开源软件,可以用于处理代谢组学profile数据,并且具有可视化和很多的分析功能。MZmine 2的一部分功能基于R,所以首先需要安装R环境,并且安装一些依赖包:instal... 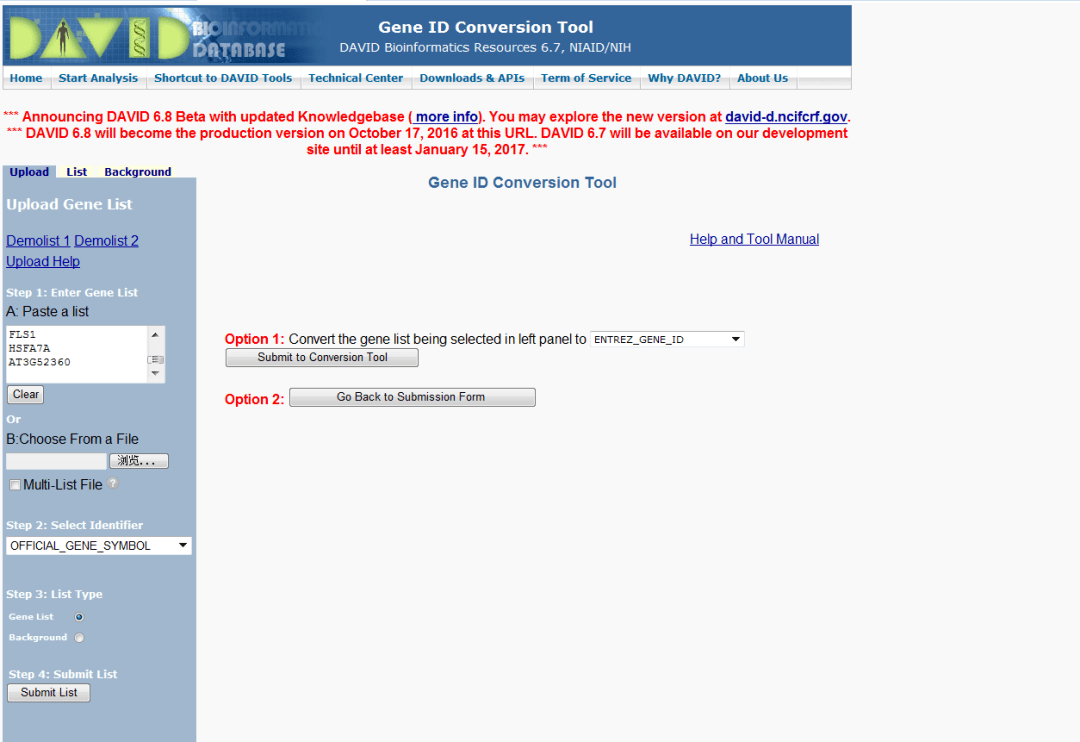 在进行差异基因表达分析时,得到显著差异基因后,接下来就需要分析这些基因参与了哪些功能,常见的就是GO功能注释和KEGG通路富集分析,今天就来介绍一下如何使用在线分析工具DAVID与KOBAS的进行KEGG通路富集分析。操作步骤进入DAVIDhttps://david.ncifcrf.gov/conversion.jsp上传基因列表选择物种开始分析选择转换选择格式,按照ENTREZ_GENE_ID转...
在进行差异基因表达分析时,得到显著差异基因后,接下来就需要分析这些基因参与了哪些功能,常见的就是GO功能注释和KEGG通路富集分析,今天就来介绍一下如何使用在线分析工具DAVID与KOBAS的进行KEGG通路富集分析。操作步骤进入DAVIDhttps://david.ncifcrf.gov/conversion.jsp上传基因列表选择物种开始分析选择转换选择格式,按照ENTREZ_GENE_ID转... 